5 Функциональное программирование в R
5.1 Создание функций
Поздравляю, сейчас мы выйдем на качественно новый уровень владения R. Вместо того, чтобы пользоваться теми функциями, которые уже написали за нас, мы можем сами создавать свои функции! В этом нет ничего сложного.
Синтаксис создания функции внешне похож на создание циклов или условных конструкций. Мы пишем ключевое слово function, в круглых скобках обозначаем переменные, с которыми собираемся что-то делать. Внутри фигурных скобок пишем выражения, которые будут выполняться при запуске функции. У функции есть свое собственное окружение — место, где хранятся переменные. Именно те объекты, которые мы передаем в скобочках, и будут в окружении, так же как и “обычные” переменные для нас в глобальном окружении. Это означает, что функция будет искать переменные в первую очередь среди объектов, которые переданы в круглых скобочках. С ними функция и будет работать. На выходе функция выдаст то, что вычисляется внутри функции return(). Если return() появляется в теле функции несколько раз, то до результат будет возвращаться из той функции return(), до которой выполнение дошло первым.
pow <- function(x, p) {
power <- x ^ p
return(power)
}
pow(3, 2)## [1] 9Если функция проработала до конца, а функция return() так и не встретилась, то возвращается последнее посчитанное значение.
pow <- function(x, p) {
x ^ p
}
pow(3, 2)## [1] 9Если в последней строчке будет присвоение, то функция ничего не вернет обратно. Это очень распространенная ошибка: функция вроде бы работает правильно, но ничего не возвращает. Нужно писать так, как будто бы в последней строчке результат выполнения выводится в консоль.
pow <- function(x, p) {
power <- x ^ p #Функция ничего не вернет, потому что в последней строчке присвоение!
}
pow(3, 2) #ничего не возвращается из функцииЕсли функция небольшая, то ее можно записать в одну строчку без фигурных скобок.
pow <- function(x, p) x ^ p
pow(3, 2) ## [1] 9Вообще, фигурные скобки используются для того, чтобы выполнить серию выражений, но вернуть только результат выполнения последнего выражения. Это можно использовать, чтобы не создавать лишних временных переменных в глобальном окружении.
Мы можем оставить в функции параметры по умолчанию.
pow <- function(x, p = 2) x ^ p
pow(3) ## [1] 9pow(3, 3) ## [1] 27В R работают ленивые вычисления (lazy evaluations). Это означает, что параметры функций будут только когда они понадобятся, а не заранее. Например, эта функция не будет выдавать ошибку, если мы не зададим параметр we_will_not_use_this_parameter =, потому что он нигде не используется в расчетах.
pow <- function(x, p = 2, we_will_not_use_this_parameter) x ^ p
pow(x = 3)## [1] 95.2 Проверка на адекватность
Лучший способ не бояться ошибок и предупреждений — научиться прописывать их самостоятельно в собственных функциях. Это позволит понять, что за текстом предупреждений и ошибок, которые у вас возникают, стоит забота разработчиков о пользователях, которые хотят максимально обезопасить нас от наших непродуманных действий.
Хорошо написанные функции не только выдают правильный результат на все возможные адекватные данные на входе, но и не дают получить правдоподобные результаты при неадекватных входных данных. Как вы уже знаете, если на входе у вас имеются пропущенные значения, то многие функции будут в ответ тоже выдавать пропущенные значения. И это вполне осознанное решение, которое позволяет избегать ситуаций вроде той, когда около одной пятой научных статей по генетике содержало ошибки в приложенных данных и замечать пропущенные значения на ранней стадии. Кроме того, можно проводить проверки на адекватность входящих данных (sanity check).
Разберем это на примере самодельной функции imt(), которая выдает индекс массы тела, если на входе задать вес (аргумент weight =) в килограммах и рост (аргумент height =) в метрах.
imt <- function(weight, height) weight / height ^ 2Проверим, что функция работает верно:
w <- c(60, 80, 120)
h <- c(1.6, 1.7, 1.8)
imt(weight = w, height = h)## [1] 23.43750 27.68166 37.03704Очень легко перепутать и написать рост в сантиметрах. Было бы здорово предупредить об этом пользователя, показав ему предупреждающее сообщение, если рост больше, чем, например, 3. Это можно сделать с помощью функции warning()
imt <- function(weight, height) {
if (height > 3) warning("Рост в аргументе height больше 3: возможно, указан рост в сантиметрах, а не в метрах\n")
weight / height ^ 2
}
imt(78, 167)## Warning in imt(78, 167): Рост в аргументе height больше 3: возможно, указан рост в сантиметрах, а не в метрах## [1] 0.002796802В некоторых случаях ответ будет совершенно точно некорректным, хотя функция все посчитает и выдаст ответ, как будто так и надо. Например, если какой-то из аргументов функции imt() будет меньше или равен 0. В этом случае нужно прописать проверку на это условие, и если это действительно так, то выдать пользователю ошибку.
imt <- function(weight, height) {
if (any(weight <= 0 | height <= 0)) stop("Индекс массы тела не может быть посчитан для отрицательных значений")
if (height > 3) warning("Рост в аргументе height больше 3: возможно, указан рост в сантиметрах, а не в метрах\n")
weight / height ^ 2
}
imt(-78, 167)## Error in imt(-78, 167): Индекс массы тела не может быть посчитан для отрицательных значенийКогда вы попробуете самостоятельно прописывать предупреждения и ошибки в функциях, то быстро поймете, что ошибки - это вовсе не обязательно результат того, что где-то что-то сломалось и нужно паниковать. Совсем даже наоборот, прописанная ошибка - чья-то забота о пользователях, которых пытаются максимально проинформировать о том, что и почему пошло не так.
Это естественно в начале работы с R (и вообще с программированием) избегать ошибок, конечно, в самом начале обучения большая часть из них остается непонятной. Но постарайтесь понять текст ошибки, вспомнить в каких случаях у вас возникала похожая ошибка. Очень часто этого оказывается достаточно чтобы понять причину ошибки даже если вы только-только начали изучать R.
Ну а в дальнейшем я советую ознакомиться со средствами отладки кода в R для того, чтобы научиться справляться с ошибками в своем коде на более продвинутом уровне.
5.3 Когда и зачем создавать функции?
Когда стоит создавать функции? Существует “правило трех” — если у вас есть три куска очень похожего кода, то самое время превратить код в функцию. Это очень условное правило, но, действительно, стоит избегать копипастинга в коде. В этом случае очень легко ошибиться, а сам код становится нечитаемым.
Есть и другой подход к созданию функций: их стоит создавать не столько для того, чтобы использовать тот же код снова, сколько для абстрагирования от того, что происходит в отдельных строчках кода. Если несколько строчек кода были написаны для того, чтобы решить одну задачу, которой можно дать понятное название (например, подсчет какой-то особенной метрики, для которой нет готовой функции в R), то этот код стоит обернуть в функцию. Если функция работает корректно, то теперь не нужно думать над тем, что происходит внутри нее. Вы ее можете мысленно представить как операцию, которая имеет определенный вход и выход — как и встроенные функции в R.
Отсюда следует важный вывод, что хорошее название для функции — это очень важно. Очень, очень, очень важно.
5.4 Функции как объекты первого порядка
Ранее мы убедились, что арифметические операторы — это тоже функции. На самом деле, практически все в R — это функции. Даже function — это функция function(). Даже скобочки (, { — это функции!
А сами функции — это объекты первого порядка в R. Это означает, что с функциями вы можете делать практически все то же самое, что и с другими объектами в R (векторами, датафреймами и т.д.). Небольшой пример, который может взорвать ваш мозг:
list(mean, min, `{`)## [[1]]
## function (x, ...)
## UseMethod("mean")
## <bytecode: 0x55b0a9afcd90>
## <environment: namespace:base>
##
## [[2]]
## function (..., na.rm = FALSE) .Primitive("min")
##
## [[3]]
## .Primitive("{")Мы можем создать список из функций! Зачем — это другой вопрос, но ведь можем же!
Еще можно создавать функции внутри функций,10 использовать функции в качестве аргументов функций, сохранять функции как переменные. Пожалуй, самое важное из этого всего - это то, что функция может быть аргументом в функции. Не просто название функции как строковая переменная, не результат выполнения функции, а именно сама функция. Это лежит в основе использования семейства функций apply() (@ref(apply_f) и многих фишек tidyverse.
В Python дело обстоит похожим образом: функции там тоже являются объектами первого порядка, поэтому все эти фишки функционального программирования (с поправкой на синтаксис, конечно) будут работать и там.
5.5 Семейство функций apply()
5.5.1 Применение apply() для матриц
Семейство? Да, их целое множество: apply(), lapply(),sapply(), vapply(),tapply(),mapply(), rapply()… Ладно, не пугайтесь, всех их знать не придется. Обычно достаточно первых двух-трех. Проще всего пояснить как они работают на простой матрице с числами:
A <- matrix(1:12, 3, 4)
A ## [,1] [,2] [,3] [,4]
## [1,] 1 4 7 10
## [2,] 2 5 8 11
## [3,] 3 6 9 12Функция
apply()предназначена для работы с матрицами (или многомерными массивами). Если вы скормите функцииapply()датафрейм, то этот датафрейм будет сначала превращен в матрицу. Главное отличие матрицы от датафрейма в том, что в матрице все значения одного типа, поэтому будьте готовы, что сработает имплицитное приведение к общему типу данных. Например, если среди колонок датафрейма есть хотя бы одна строковая колонка, то все колонки станут строковыми.
Теперь представим, что нам нужно посчитать что-нибудь (например, сумму) по каждой из строк. С помощью функции apply() вы можете в буквальном смысле “применить” функцию к матрице или датафрейму. Синтаксис такой: apply(X, MARGIN, FUN, ...), где X — данные, MARGIN это 1 (для строк), 2 (для колонок), c(1,2) для строк и колонок (т.е. для каждого элемента по отдельности), а FUN — это функция, которую вы хотите применить! apply() будет брать строки/колонки из X в качестве первого аргумента для функции.
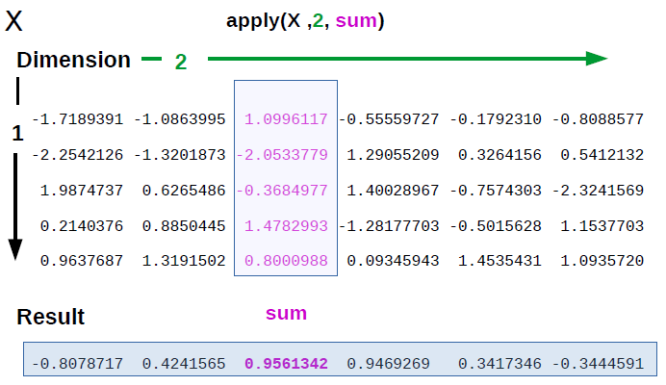
apply
Заметьте, мы вставляем функцию без скобок и кавычек как аргумент в функцию. Это как раз тот случай, когда аргументом в функции выступает сама функция, а не ее название или результат ее выполнения.
Давайте разберем на примере:
apply(A, 1, sum) #сумма по каждой строчке## [1] 22 26 30apply(A, 2, sum) #сумма по каждой колонке## [1] 6 15 24 33apply(A, c(1,2), sum) #кхм... сумма каждого элемента## [,1] [,2] [,3] [,4]
## [1,] 1 4 7 10
## [2,] 2 5 8 11
## [3,] 3 6 9 12Конкретно для подсчета сумм и средних по столбцам и строкам в R есть функции
colSums(),rowSums(),colMeans()иrowMeans(), которые можно использовать как альтернативыapply()в данном случае.
Если же мы хотим прописать дополнительные аргументы для функции, то их можно перечислить через запятую после функции:
apply(A, 1, sum, na.rm = TRUE)## [1] 22 26 30apply(A, 1, weighted.mean, w = c(0.2, 0.4, 0.3, 0.1)) ## [1] 4.9 5.9 6.95.5.2 Анонимные функции
Что делать, если мы хотим сделать что-то более сложное, чем просто применить одну функцию? А если функция принимает не первым, а вторым аргументом данные из матрицы? В этом случае нам помогут анонимные функции.
Анонимные функции - это функции, которые будут использоваться один раз и без названия.
Питонистам знакомо понятие лямбда-функций. Да, это то же самое.
Например, мы можем посчитать отклонения от среднего без называния этой функции:
apply(A, 1, function(x) x - mean(x)) #отклонения от среднего по строчке## [,1] [,2] [,3]
## [1,] -4.5 -4.5 -4.5
## [2,] -1.5 -1.5 -1.5
## [3,] 1.5 1.5 1.5
## [4,] 4.5 4.5 4.5apply(A, 2, function(x) x - mean(x)) #отклонения от среднего по столбцу## [,1] [,2] [,3] [,4]
## [1,] -1 -1 -1 -1
## [2,] 0 0 0 0
## [3,] 1 1 1 1apply(A, c(1,2), function(x) x - mean(x)) #отклонения от одного значения, т.е. ноль## [,1] [,2] [,3] [,4]
## [1,] 0 0 0 0
## [2,] 0 0 0 0
## [3,] 0 0 0 0Как и в случае с обычной функцией, в качестве x выступает объект, с которым мы хотим что-то сделать, а дальше следует функция, которую мы собираемся применить к х. Можно использовать не х, а что угодно, как и в обычных функциях:
apply(A, 1, function(whatevername) whatevername - mean(whatevername))## [,1] [,2] [,3]
## [1,] -4.5 -4.5 -4.5
## [2,] -1.5 -1.5 -1.5
## [3,] 1.5 1.5 1.5
## [4,] 4.5 4.5 4.55.5.3 Другие функции семейства apply()
Ок, с apply() разобрались. А что с остальными? Некоторые из них еще проще и не требуют индексов, например, lapply (для применения к каждому элементу списка) и sapply() - упрощенная версия lapply(), которая пытается по возможности “упростить” результат до вектора или матрицы.
some_list <- list(some = 1:10, list = letters)
lapply(some_list, length)## $some
## [1] 10
##
## $list
## [1] 26sapply(some_list, length)## some list
## 10 26Использование sapply() на векторе приводит к тем же результатам, что и просто применить векторизованную функцию обычным способом.
sapply(1:10, sqrt)## [1] 1.000000 1.414214 1.732051 2.000000 2.236068 2.449490 2.645751 2.828427
## [9] 3.000000 3.162278sqrt(1:10)## [1] 1.000000 1.414214 1.732051 2.000000 2.236068 2.449490 2.645751 2.828427
## [9] 3.000000 3.162278Зачем вообще тогда нужен sapply(), если мы можем просто применить векторизованную функцию? Ключевое слово здесь векторизованная функция. Если функция не векторизована, то sapply() становится удобным вариантом для того, чтобы избежать итерирования с помощью циклов for.
Еще одна альтернатива - это векторизация невекторизованной функции с помощью
Vectorize(). Эта функция просто оборачивает функцию одним из вариантовapply().
Можно применять функции lapply() и sapply() на датафреймах. Поскольку фактически датафрейм - это список из векторов одинаковой длины (см. 2.11), то итерироваться эти функции будут по колонкам:
heroes <- read.csv("data/heroes_information.csv",
na.strings = c("-", "-99"))
sapply(heroes, class)## X name Gender Eye.color Race Hair.color
## "integer" "character" "character" "character" "character" "character"
## Height Publisher Skin.color Alignment Weight
## "numeric" "character" "character" "character" "integer"Еще одна функция из семейства apply() - функция replicate() - самый простой способ повторить одну и ту же операцию много раз. Обычно эта функция используется при симуляции данных и моделировании. Например, давайте сделаем выборку из логнормального распределения:
samp <- rlnorm(30)
hist(samp)
А теперь давайте сделаем 1000 таких выборок и из каждой возьмем среднее:
sampdist <- replicate(1000, mean(rlnorm(30)))
hist(sampdist)
Про функции для генерации случайных чисел и про визуализацию мы поговорим в следующие дни.
Если хотите познакомиться с семейством apply() чуточку ближе, то рекомендую вот этот туториал.
В заключение стоит сказать, что семейство функций apply() — это очень сильное колдунство, но в tidyverse оно практически полностью перекрывается функциями из пакета purrr. Впрочем, если вы поняли логику apply(), то при желании вы легко сможете переключиться на альтернативы из пакета purrr.
#Введение в tidyverse {#tidy_intro}
5.6 Вселенная tidyverse
tidyverse - это не один, а целое множество пакетов. Есть ключевые пакеты (ядро тайдиверса), а есть побочные - в основном для работы со специфическими видами данных.
tidyverse — это набор пакетов:
- ggplot2, для визуализации
- tibble, для работы с тибблами, продвинутый вариант датафрейма
- tidyr, для формата tidy data
- readr, для чтения файлов в R
- purrr, для функционального программирования (замена семейства функций *apply())
- dplyr, для преобразованиия данных
- stringr, для работы со строковыми переменными
- forcats, для работы с переменными-факторами
Полезно также знать о следующих пакетах, не включенных в ядро, но также считающихся частью тайдиверса:
- vroom, для быстрой загрузки табоичных данных
- readxl, для чтения .xls и .xlsx
- jsonlite, для работы с JSON
- xml, для работы с XML
- DBI, для работы с базами данных
- rvest, для веб-скреппинга
- lubridate, для работы с временем
- tidytext, для работы с текстами и корпусами
- glue, для продвинутого объединения строк
- magrtittr, с несколькими вариантами pipe оператора
- tidymodels, для моделирования и машинного обучения11
- dtplyr, для ускорения
dplyrза счет перевод синтаксиса наdata.table
И это еще не все пакеты tidyverse! Есть еще много других небольших пакетов, которые тоже считаются частью tidyverse. Кроме официальных пакетов tidyverse есть множество пакетов, которые пытаются соответствовать принципам tidyverse и дополняют его.
Все пакеты tidyverse объединены tidy философией и взаимосовместимым синтаксисом. Это означает, что, во многих случаях даже не нужно думать о том, из какого именно пакета тайдиверса пришла функция. Можно просто установить и загрузить пакет tidyverse.
install.packages("tidyverse")Пакет tidyverse — это такой пакет с пакетами.
library("tidyverse")## ── Attaching packages ─────────────────────────────────────── tidyverse 1.3.1 ──## ✓ ggplot2 3.3.3 ✓ purrr 0.3.4
## ✓ tibble 3.1.2 ✓ dplyr 1.0.6
## ✓ tidyr 1.1.3 ✓ stringr 1.4.0
## ✓ readr 1.4.0 ✓ forcats 0.5.1## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()Подключение пакета tidyverse автоматически приводит к подключению ядра tidyverse, остальные же пакеты нужно подключать дополнительно при необходимости.
5.7 Загрузка данных с помощью readr
Стандартной функцией для чтения .csv файлов в R является функция read.csv(), но мы будем использовать функцию read_csv() из пакета readr. Синтаксис функции read_csv() очень похож на read.csv(): первым аргументом является путь к файлу (в том числе можно использовать URL), некоторые остальные параметры тоже совпадают.
heroes <- read_csv("data/heroes_information.csv",
na = c("-", "-99"))## Warning: Missing column names filled in: 'X1' [1]##
## ── Column specification ────────────────────────────────────────────────────────
## cols(
## X1 = col_double(),
## name = col_character(),
## Gender = col_character(),
## `Eye color` = col_character(),
## Race = col_character(),
## `Hair color` = col_character(),
## Height = col_double(),
## Publisher = col_character(),
## `Skin color` = col_character(),
## Alignment = col_character(),
## Weight = col_double()
## )Подробнее про импорт данных, в том числе в tidyverse, смотри в @ref(real_data).
##tibble
Когда мы загрузили данные с помощью read_csv(), то мы получили tibble, а не data.frame:
class(heroes)## [1] "spec_tbl_df" "tbl_df" "tbl" "data.frame"Тиббл (tibble) - это такой “усовершенствованный” data.frame. Почти все, что работает с data.frame, работает и с тибблами. Однако у тибблов есть свои дополнительные фишки. Самая очевидная из них - более аккуратный вывод в консоль:
heroesВыводятся только первые 10 строк, если какие-то колонки не влезают на экран, то они просто перечислены внизу. Ну а тип данных написан прямо под названием колонки.
Функции различных пакетов tidyverse сами конвертируют в тиббл при необходимости. Если же нужно это сделать самостоятельно, то можно это сделать так:
heroes_df <- as.data.frame(heroes) #создаем простой датафрейм
class(heroes_df)## [1] "data.frame"as_tibble(heroes_df) #превращаем обратно в тибблВ дальнейшем мы будем работать только с tidyverse, а это значит, что только с тибблами, а не обычными датафреймами. Тем не менее, тибблы и датафреймы будут в дальнейшем использоваться как синонимы.
Можно создавать тибблы вручную с помощью функции tibble(), которая работает аналогично функции data.frame():
tibble(
a = 1:3,
b = letters[1:3]
)5.8 magrittr::%>%
Оператор %>% называется “пайпом” (pipe), т.е. “трубой.” Он означает, что следующая функция (справа от пайпа) принимает на вход в качестве первого аргумента результат выполнения предыдущей функции (той, что слева). Фактически, это примерно то же самое, что и вставлять результат выполнения функции в качестве первого аргумента в другую функцию. Просто выглядит это красивее и читабельнее. Как будто данные пропускаются через трубы функций или конвеерную ленту на заводе, если хотите. А то, что первый параметр функции - это почти всегда данные, работает нам здесь на руку. Этот оператор взят из пакета magrittr12. Возможно, даже если вы не захотите пользоваться tidyverse, использование пайпов Вам понравится.
Важно понимать, что пайп не дает какой-то дополнительной функциональности или дополнительной скорости работы13. Он создан исключительно для читабельности и комфорта.
С помощью пайпов вот эту команду…
sum(sqrt(abs(sin(1:22))))## [1] 16.72656…можно переписать вот так:
1:22 %>%
sin() %>%
abs() %>%
sqrt() %>%
sum()## [1] 16.72656В очень редких случаях результат выполнения функции нужно вставить не на первую позицию (или же мы хотим использовать его несколько раз). В этих случаях можно использовать ., чтобы обозначить, куда мы хотим вставить результат выполнения выражения слева от %>%.
"Всем привет!" %>%
c("--", ., "--")## [1] "--" "Всем привет!" "--"5.9 Главные пакеты tidyverse: dplyr и tidyr
dplyr14 — это самая основа всего tidyverse. Этот пакет предоставляет основные функции для манипуляции с тибблами. Пакет dplyr является наследником и более усовершенствованной версией plyr, так что если увидите использование пакета plyr, то, скорее всего, скрипт был написан очень давно.
Пакет tidyr дополняет dplyr, предоставляя полезные функции для тайдификации тибблов.
Тайдификация (“аккуратизация”) данных означает приведение табличных данных к такому формату, в котором:
- Каждая переменная имеет собственный столбец
- Каждый наблюдение имеет собственную строку
- Каждое значение имеет свою собственную ячейку
Впрочем, многие функции dplyr часто используются при тайдификации, так же как и многие функции tidyr имеет применение вне тайдификации. В общем, функционал этих двух пакетов несколько смешался, поэтому мы будем рассматривать их вместе. А чтобы представлять, какая функция относится к какому пакету (хотя запоминать это необязательно), я буду использовать запись с двумя двоеточиями ::, которая обычно используется для использования функции без подгрузки всего пакета, при первом упоминании функции.
Пакет tidyr — это более усовершенствованная версия пакета reshape2, который в свою очередь является усовершенствованной версией reshape. По аналогии с plyr, если вы видите использование этих пакетов, то это указывает на то, что перед вами морально устаревший код.
Код с использованием dplyr и tidyrсильно непохож на то, что мы видели раньше. Большинство функций dplyr и tidyr работают с целым тибблом сразу, принимая его в качестве первого аргумента и возвращая измененный тиббл. Это позволяет превратить весь код в последовательный набор применяемых функций, соединенный пайпами. На практике это выглядит очень элегантно, и вы в этом скоро убедитесь.
5.10 Работа с колонками тиббла
5.10.1 Выбор колонок: dplyr::select()
Функция dplyr::select() позволяет выбирать колонки по номеру или имени (кавычки не нужны).
heroes %>%
select(1,5)heroes %>%
select(name, Race, Publisher, `Hair color`)Обратите внимание, если в названии колонки присутствует пробел или, например, колонка начинается с цифры или точки и цифры, то это синтаксически невалидное имя (2.5.3). Это не значит, что такие названия колонок недопустимы. Но такие названия колонок нужно обособлять ` грависом (правый штрих, на клавиатуре находится там же где и буква ё и ~).
Еще обратите внимание на то, что функции tidyverse не изменяют сами изначальные тибблы/датафреймы. Это означает, что если вы хотите полученный результат сохранить, то нужно добавить присвоение:
heroes_some_cols <- heroes %>%
select(name, Race, Publisher, `Hair color`)
heroes_some_cols5.10.2 Мини-язык tidyselect для выбора колонок
Для выбора столбцов (не только в select(), но и для других функций tidyverse) используется специальный мини-язык tidyselect из одноименного пакета15. tidyselect дает очень широкие возможности для выбора колонок.
Можно использовать оператор : для выбора нескольких соседних колонок (по аналогии с созданием числового вектора с шагом 1).
heroes %>%
select(name:Publisher)heroes %>%
select(name:`Eye color`, Publisher:Weight)Используя ! можно вырезать ненужные колонки.
heroes %>%
select(!X1)heroes %>%
select(!(Gender:Height))Другие известные нам логические операторы (& и |) тоже работают в tidyselect.
В дополнение к логическим операторам и :, в tidyselect есть набор вспомогательных функций, работающих исключительно в контексте выбора колонок с помощью tidyselect.
Вспомогательная функция last_col() позволит обратиться к последней колонке тиббла:
heroes %>%
select(name:last_col())А функция everything() позволяет выбрать все колонки.
heroes %>%
select(everything())При этом everything() не будет дублировать выбранные колонки, поэтому можно использовать everything() для перестановки колонок в тиббле:
heroes %>%
select(name, Publisher, everything())Впрочем, для перестановки колонок удобнее использовать специальную функцию relocate() (@ref(tidy_relocate))
Можно даже выбирать колонки по паттернам в названиях. Например, с помощью ends_with() можно выбрать все колонки, заканчивающиеся одинаковым суффиксом:
heroes %>%
select(ends_with("color"))Аналогично, с помощью функции starts_with() можно найти колонки с одинаковым префиксом, с помощью contains() — все колонки с выбранным паттерном в любой части названия колонки16.
heroes %>%
select(starts_with("Eye") & ends_with("color"))heroes %>%
select(contains("eight"))Ну и наконец, можно выбирать по содержимому колонок с помощью where(). Это напоминает применение sapply()(@ref(apply_other)) на датафрейме для индексирования колонок: в качестве аргумента для where принимается функция, которая применяется для каждой из колонок, после чего выбираются только те колонки, для которых было получено TRUE.
heroes %>%
select(where(is.numeric))Функция where() дает невиданную мощь. Например, можно выбрать все колонки без NA:
heroes %>%
select(where(function(x) !any(is.na(x))))###Переименование колонок: dplyr::rename()
Внутри select() можно не только выбирать колонки, но и переименовывать их:
heroes %>%
select(id = X1)Однако удобнее для этого использовать специальную функцию dplyr::rename(). Синтаксис у нее такой же, как и у select(), но rename() не выбрасывает колонки, которые не были упомянуты.
heroes %>%
rename(id = X1)Для массового переименования колонок можно использовать функцию rename_with(). Эта функция так же использует tidyselect синтаксис для выбора колонок (по умолчанию выбираются все колонки) и применяет функцию в качестве аргумента, которая изменяет
heroes %>%
rename_with(make.names)###Перестановка колонок: dplyr::relocate() {#tidy_relocate}
Для изменения порядка колонок можно использовать функцию relocate(). Она тоже работает похожим образом на select() и rename()17. Как и rename(), функция relocate() не выкидывает неиспользованные колонки:
heroes %>%
relocate(Publisher)При этом relocate() имеет дополнительные параметры .after = и .before =, которые позволяют выбирать, куда поместить выбранные колонки.
heroes %>%
relocate(Publisher, .after = name)relocate() очень хорошо работает в сочетании с выбором колонок с помощью tidyselect. Например, можно передвинуть в одно место все колонки с одним типом данных:
heroes %>%
relocate(Publisher, where(is.numeric), .after = name)Последняя важная функция для выбора колонок — pull(). Эта функция делает то же самое, что и индексирование с помощью $, т.е. вытаскивает из тиббла вектор с выбранным названием. Это лучше вписывается в логику tidyverse, поскольку позволяет извлечь колонку из тиббла с использованием пайпа:
heroes %>%
select(Height) %>%
pull() %>%
head()## [1] 203 191 185 203 NA 193heroes %>%
pull(Height) %>%
head()## [1] 203 191 185 203 NA 193У функции pull() есть аргумент name =, который позволяет создать проименованный вектор:
heroes %>%
pull(Height, name) %>%
head()## A-Bomb Abe Sapien Abin Sur Abomination Abraxas
## 203 191 185 203 NA
## Absorbing Man
## 193В отличие от базового R, tidyverse нигде не сокращает имплицитно результат вычислений до вектора, поэтому функция pull() - это основной способ извлечения колонки из тиббла как вектора.
5.11 Работа со строками тиббла
5.11.1 Выбор строк по номеру: dplyr::slice()
Начнем с выбора строк. Функция dplyr::slice() выбирает строчки по их числовому индексу.
heroes %>%
slice(1:3)5.11.2 Выбор строк по условию: dplyr::filter()
Функция dplyr::filter() делает то же самое, что и slice(), но уже по условию. Причем для условий нужно использовать не векторы из тиббла, а название колонок (без кавычек) как будто бы они были переменными в окружении.
heroes %>%
filter(Publisher == "DC Comics")5.11.3 Семейство функций slice()
У функции slice() есть множество родственников, которые объединяют функционал обычного slice() и filter(). Например, с помощью функций dplyr::slice_max() и dplyr::slice_min() можно выбрать заданное количество строк, содержащих наибольшие или наименьшие значения по колонке соответственно:
heroes %>%
slice_max(Weight, n = 3)heroes %>%
slice_min(Weight, n = 3)Функция slice_sample() позволяет выбирать заданное количество случайных строчек:
heroes %>%
slice_sample(n = 3)Или же долю строчек:
heroes %>%
slice_sample(prop = .01)Если поставить значение параметра prop = равным 1, то таким образом можно перемешать порядок строчек в тиббле:
heroes %>%
slice_sample(prop = 1)5.11.4 Удаление строчек с NA: tidyr::drop_na()
Если нужно выбрать только строчки без пропущенных значений, то можно воспользоваться удобной функцией tidyr::drop_na().
heroes %>%
drop_na()Можно выбрать колонки, наличие NA в которых будет приводить к удалению соответствующих строчек (не затрагивая другие строчки, в которых есть NA в остальных столбцах).
heroes %>%
drop_na(Weight)Для выбора колонок в drop_na() используется tidyselect, с которым мы недавно познакомились (5.10.2).
5.11.5 Сортировка строк: dplyr::arrange()
Функция dplyr::arrange() сортирует строчки от меньшего к большему (или по алфавиту - для текстовых значений) по выбранной колонке.
heroes %>%
arrange(Weight)Чтобы отсортировать в обратном порядке, воспользуйтесь функцией desc().
heroes %>%
arrange(desc(Weight))Можно сортировать по нескольким колонкам сразу. В таких случаях удобно в качестве первой переменной выбирать переменную, обозначающую принадлежность к группе, а в качестве второй — континуальную числовую переменную:
heroes %>%
arrange(Gender, desc(Weight))5.12 Создание колонок: dplyr::mutate() и dplyr::transmute()
Функция dplyr::mutate() позволяет создавать новые колонки в тиббле.
heroes %>%
mutate(imt = Weight/(Height/100)^2) %>%
select(name, imt) %>%
arrange(desc(imt))dplyr::transmute() - это аналог mutate(), который не только создает новые колонки, но и сразу же выкидывает все старые:
heroes %>%
transmute(imt = Weight/(Height/100)^2)Внутри mutate() и transmute() мы можем использовать либо векторизованные операции (длина новой колонки должна равняться длине датафрейма), либо операции, которые возвращают одно значение. В последнем случае значение будет одинаковым на всю колонку, т.е. будет работать правило ресайклинга (2.8.3):
heroes %>%
transmute(name, weight_mean = mean(Weight, na.rm = TRUE))Однако в функциях mutate() и transmute() правило ресайклинга не будет работать в остальных случаях: если полученный вектор будет не равен 1 или длине датафрейма, то мы получим ошибку.
heroes %>%
mutate(one_and_two = 1:2)## Error: Problem with `mutate()` column `one_and_two`.
## ℹ `one_and_two = 1:2`.
## ℹ `one_and_two` must be size 734 or 1, not 2.Это не баг, а фича: авторы пакета dplyr считают, что ресайклинг кратных друг другу векторов — это слишком удобное место для выстрелов себе в ногу. Поэтому в таких случаях разработчики dplyr рекомендуют использовать функцию rep(), знакомую нам уже очень давно (2.8).
heroes %>%
mutate(one_and_two = rep(1:2, length.out = nrow(.)))5.13 Агрегация данных в тиббле
5.13.1 Подытоживание: summarise()
Аггрегация по группам - это очень часто возникающая задача, например, это может использоваться для усреднения данных по испытуемым или условиям. Сделать аггрегацию в датафрейме удобной Хэдли Уикхэм пытался еще в предшественнике dplyr, пакете plyr. dplyr позволяет делать аггрегацию очень симпатичным и понятным способым. Аггрегация в dplyr состоит из двух этапов: группировки (group_by()) и подытоживания (summarise()). Начнем с последнего.
Функция dplyr::summarise()18 позволяет аггрегировать данные в тиббле. Работает она очень похоже на mutate(), но если внутри mutate() используются векторизованные функции, возвращающие вектор такой же длины, что и колонки, использовавшиеся для расчетов, то в summarise() используются функции, которые возвращают вектор длиной 1. Например, min(), mean(), max() и т.д. Можно создавать несколько колонок через запятую (это работает и для mutate()).
heroes %>%
mutate(imt = Weight/(Height/100)^2) %>%
summarise(min(imt, na.rm = TRUE),
max(imt, na.rm = TRUE))В dplyr есть дополнительные суммирующие функции для более удобного индексирования в стиле tidyverse. Например, функции dplyr::nth(), dplyr::first() и dplyr::last(), которые позволяют вытаскивать значения из вектора по индексу (что-то вроде slice(), но для векторов)
heroes %>%
mutate(imt = Weight/(Height/100)^2) %>%
arrange(imt) %>%
summarise(first = first(imt),
tenth = nth(imt, 10),
last = last(imt))В отличие от mutate(), функции внутри summarise() вполне позволяют функциям внутри возвращать вектор из нескольких значений, создавая тиббл такой же длины, как и получившийся вектор.
heroes %>%
mutate(imt = Weight/(Height/100)^2) %>%
summarise(imt_range = range(imt, na.rm = TRUE)) #функция range() возвращает вектор из двух значений: минимальное и максимальное5.13.2 Группировка: group_by()
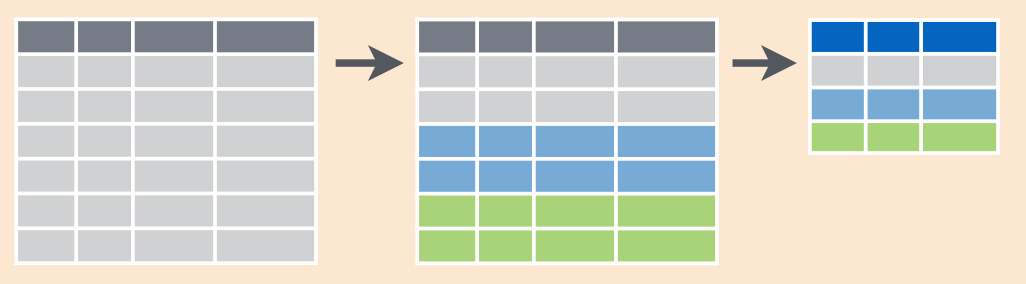
dplyr::group_by() - это функция для группировки данных в тиббле по дискретной переменной для дальнейшей аггрегации с помощью summarise(). После применения group_by() тиббл будет выглядеть так же, но у него появятся атрибут groups19:
heroes %>%
group_by(Gender)Если после этого применить на тиббле функцию summarise(), то мы получим не тиббл длиной один, а тиббл со значением для каждой из групп.
heroes %>%
mutate(imt = Weight/(Height/100)^2) %>%
group_by(Gender) %>%
summarise(min(imt, na.rm = TRUE),
max(imt, na.rm = TRUE))Схематически это выглядит вот так:
5.13.3 Подсчет строк: dplyr::n(), dplyr::count()
Для подсчет количества значений можно воспользоваться функцией n().
heroes %>%
group_by(Gender) %>%
summarise(n = n())Функция n() вместе с group_by() внутри filter() позволяет удобным образом “отрезать” от тиббла редкие группы…
heroes %>%
group_by(Race) %>%
filter(n() > 10) %>%
select(name, Race)или же наоборот, выделить только маленькие группы:
heroes %>%
group_by(Race) %>%
filter(n() == 1) %>%
select(name, Race)Таблицу частот можно создать без group_by() и summarise(n = n()). Функция count() заменяет эту конструкцию:
heroes %>%
count(Gender)Эту таблицу частот удобно сразу проранжировать, указав в параметре sort = значение TRUE.
heroes %>%
count(Gender, sort = TRUE)Функция
count(), несмотря на свою простоту, является одной из наиболее используемых в tidyverse.
5.13.4 Уникальные значения: dplyr::distinct()
dplyr::distinct() - это более быстрый аналог unique(), позволяет извлекать уникальные значения для одной или нескольких колонок.
heroes %>%
distinct(Gender)heroes %>%
distinct(Gender, Race)Иногда нужно аггрегировать данные, но при этом сохранить исходную структуру тиббла. Например, нужно посчитать размер групп или посчитать средние значения по группе для последующего сравнения с индивидуальными значениями.
5.13.5 Создание колонок с группировкой
В tidyverse это можно сделать с помощью сочетания group_by() и mutate() (вместо summarise()):
heroes %>%
group_by(Race) %>%
mutate(Race_n = n()) %>%
select(Race, name, Gender, Race_n)Результаты аггрегации были записаны в отдельную колонку, при этом значения этой колонки внутри одной группы повторяются:
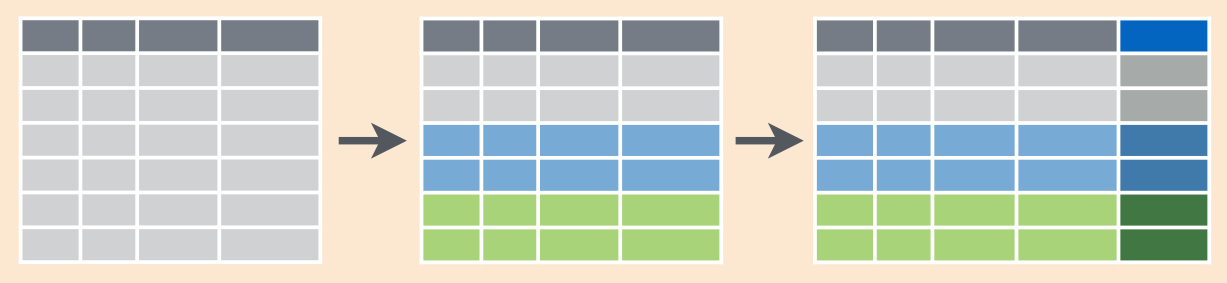
5.14 Трансформация нескольких колонок: dplyr::across()
Допустим, вы хотите посчитать среднюю массу и рост, группируя по полу супергероев. Можно посчитать это внутри одного summarise(), использую запятую:
heroes %>%
group_by(Gender) %>%
summarise(height = mean(Height, na.rm = TRUE),
weight = mean(Weight, na.rm = TRUE))Если таких колонок будет много, то это уже станет сильно неудобным, нам придется много копировать код, а это чревато ошибками и очень скучно.
Поэтому в dplyr есть функция для операций над несколькими колонками сразу: dplyr::across()20. Эта функция работает похожим образом на функции семейства apply() и использует tidyselect для выбора колонок.
Таким образом, конструкции с функцией across() можно разбить на три части:
- Выбор колонок с помощью tidyselect. Здесь работают все те приемы, которые мы изучили при выборе колонок (5.10.2).
- Собственно применение функции
across(). Первый аргумент.col— колонки, выбранные на первом этапе с помощью tidyselect, по умолчанию этоeverything(), т.е. все колонки. Второй аргумент.fns— это функция или целый список из функций, которые будут применены к выбранным колонкам. Если функции требуют дополнительных аргументов, то они могут быть перечислены внутриacross(). - Использование
summarise()или другой функцииdplyr. В этом случае в качестве аргумента для функции используется результат работы функцииacross().
Вот такой вот бутерброд выходит. Давайте посмотрим, как это работает на практике и посчитаем среднее значение по колонкам Height и Weight.
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height,Weight), mean))Здесь мы столкнулись с уже известной нам проблемой: функция mean() при столкновении хотя бы с одним NA будет возвращать NA, если мы не изменим параметр na.rm =. Как и в случае с функциями семейства apply() (@ref(apply_f)), дополнительные параметры для функции можно перечислить через запятую после самой функции:
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height, Weight), mean, na.rm = TRUE))До этого мы просто использовали выбор колонок по их названию. Но именно внутри across() использование tidyselect раскрывается как удивительно элегантный и мощный инструмент. Например, можно посчитать среднее для всех numeric колонок:
heroes %>%
drop_na(Height, Weight) %>%
group_by(Gender) %>%
summarise(across(where(is.numeric), mean, na.rm = TRUE))Или длину строк для строковых колонок. Для этого нам понадобится вспомнить, как создавать анонимные функции (@ref(anon_f)).
heroes %>%
group_by(Gender) %>%
summarise(across(where(is.character),
function(x){mean(nchar(x), na.rm = TRUE)}))Или же даже посчитать и то, и другое внутри одного summarise()!
heroes %>%
group_by(Gender) %>%
summarise(across(where(is.numeric), mean, na.rm = TRUE),
across(where(is.character),
function(x){mean(nchar(x), na.rm = TRUE)}))Внутри одного across() можно применить не одну функцию к каждой из выбранных колонок, а сразу несколько функций для каждой из колонок. Для этого нам нужно использовать список функций (желательно - проименованный).
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height, Weight),
list(minimum = min,
average = mean,
maximum = max),
na.rm = TRUE))Вот нам и понадобился список функций (@ref(functions_objects))!
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height, Weight),
list(min = function(x) min(x, na.rm = TRUE),
mean = function(x) mean(x, na.rm = TRUE),
max = function(x) max(x, na.rm = TRUE),
na_n = function(x, ...) sum(is.na(x)))
)
)Хотя основное применение функции across() — это массовое подытоживание с помощью summarise(), across() можно использовать и с другими функциями dplyr. Например, можно делать массовые операции с колонками с помощью mutate():
heroes %>%
mutate(across(where(is.character), as.factor))Менее очевидный способ применения across() - использование across() внутри count() вместе с функцией n_distinct(), которая считает количество уникальных значений в векторе. Это позволяет посмотреть таблицу частот для группирующих переменных:
heroes %>%
select(where(function(x) n_distinct(x) <= 6))heroes %>%
count(across(where(function(x) n_distinct(x) <= 6)))5.15 Объединение нескольких датафреймов
5.15.1 Соединение структурно схожих датафреймов: bind_rows(), bind_cols()
Для начала создадим следующие тибблы и сохраним их как dc, marvel и other_publishers:
dc <- heroes %>%
filter(Publisher == "DC Comics") %>%
group_by(Gender) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE))
dcmarvel <- heroes %>%
filter(Publisher == "Marvel Comics") %>%
group_by(Gender) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE))
marvelother_publishers <- heroes %>%
filter(!(Publisher %in% c("DC Comics","Marvel Comics"))) %>%
group_by(Gender) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE))
other_publishersНесколько тибблов можно объединить вертикально с помощью функции bind_rows(). Для корректного объединения тибблы должны иметь одинаковые названия колонок.
bind_rows(dc, marvel)Чтобы соединить тибблы горизонтально, воспользуйтесь функцией bind_cols().
bind_cols(dc, marvel)## New names:
## * Gender -> Gender...1
## * weight_mean -> weight_mean...2
## * Gender -> Gender...3
## * weight_mean -> weight_mean...4Функции bind_rows() и bind_cols() могут работать не только с двумя, но сразу с несколькими датафреймами.
bind_rows(dc, marvel, other_publishers)На входе в функции bind_rows() и bind_cold() можно подавать как сами датафреймы или тибблы через запятую, так и список из датафреймов/тибблов.
heroes_list_of_df <- list(DC = dc,
Marvel = marvel,
Other = other_publishers)
bind_rows(heroes_list_of_df)Чтобы не потерять, из какого датафрейма какие данные, можно указать любое строковое значение (название будущей колонки) для необязательного аргумента .id =.
bind_rows(heroes_list_of_df, .id = "Publisher")bind_rows() обычно используется, когда ваши данные находятся в разных файлах с одинаковой структурой. Тогда вы можете прочитать все таблицы в папке, сохранить их в качестве списка из датафреймов и объединить в один датафрейм с помощью bind_rows().
5.15.2 Реляционные данные: *_join()
В реальности иногда возникает ситуация, когда нужно соединить две таблички, у которых есть общий столбец (или несколько столбцов), но все остальные столбцы различаются. Табличек может быть и больше, это может быть целая сеть таблиц, некоторые из которых содержат основные данные, а некоторые - дополнительные, которые необходимо на определенном этапе “включить” в анализ. Например, таблица с расшифровкой аббревиатур или сокращений вроде коротких названий стран или таблица телефонных кодов разных стран. Совокупность нескольких связанных друг с другом таблиц называют реляционными данными.
В случае с реляционными данными простых bind_rows() и bind_cols() становится недостаточно.
Эти две таблички нужно объединить (join). Эта задача обычно возникает не очень часто, обычно это происходит один-два раза в одном проекте, когда нужно дополнить имеющиеся данные дополнительной информацией извне или объединить два набора данных, обрабатывавшихся в разных программах. Всякий раз, когда такая задача возникает, это доставляет много боли. dplyr предлагает интуитивно понятный инструмент для объединения реляционных данных - семейство функций *_join().
Возьмем для примера два тиббла band_members и band_instruments, встроенных в dplyr специально для демонстрации работы функций *_join().
band_membersband_instrumentsУ этих двух тибблов есть колонка с одинаковым названием, которая по своему смыслу соединяет данные обоих тибблов. Такая колонка называется ключом. Ключ должен однозначно идентифицировать наблюдения21.
Давайте попробуем посоединять band_members и band_instruments разными вариантами *_join() и посмотрим, что у нас получится. Все эти функции имеют на входе два обязательных аргумента (x = и y =) в которые мы должны подставить два датафрейма/тиббла которые мы хотим объединить. Главное различие между этими функциями заключается в том, что они будут делать, если уникальные значения в ключах x и y не соответствуют друг другу.
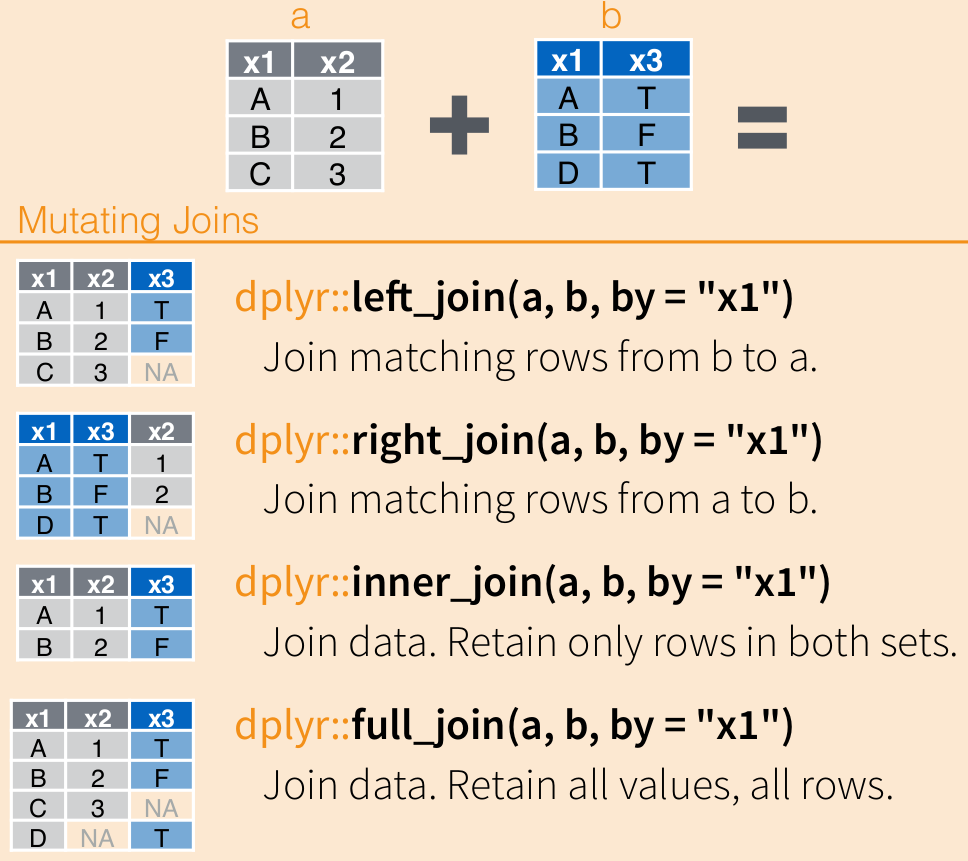
left_join():
band_members %>%
left_join(band_instruments)## Joining, by = "name"left_join() - это самая простая для понимания и самая используемая функция из семейства *_join(). Она как бы “дополняет” информацию из первого тиббла вторым тибблом. В этом случае сохраняются все уникальные наблюдения в x, но отбрасываются лишние наблюдения в тиббле y. Тем значениям, которым не нашлось соотвествия в y, в колонках, взятых их y, ставятся значения NA.
Вы можете сами задать колонки-ключи параметром by =, по умолчанию это все колонки с одинаковыми названиями в двух тибблах.
band_members %>%
left_join(band_instruments, by = "name")Часто случается, что колонки-ключи называются по-разному в двух тибблах. Их необязательно переименовывать, можно поставить соответстие вручную используя проименованный вектор:
band_members %>%
left_join(band_instruments2, by = c("name" = "artist"))right_join():
band_members %>%
right_join(band_instruments)## Joining, by = "name"right_join() отбрасывает строчки в x, которых не было в y, но сохраняет соответствующие строчки y - left_join() наоборот.
full_join():
band_members %>%
full_join(band_instruments)## Joining, by = "name"Функция full_join() сохраняет все строчки и из x и y. Пожалуй, наиболее используемая функция после left_join() — благодаря full_join() вы точно ничего не потеряете при объединении.
inner_join():
band_members %>%
inner_join(band_instruments)## Joining, by = "name"Функция full_join() сохраняет только строчки, которые присутствуют и в x, и в y.
semi_join():
band_members %>%
semi_join(band_instruments)## Joining, by = "name"anti_join():
band_members %>%
anti_join(band_instruments)## Joining, by = "name"Функции semi_join() и anti_join() не присоединяют второй датафрейм/тиббл (y) к первому. Вместо этого они используются как некоторый словарь-фильтр для отделения только тех значений в x, которые есть в y (semi_join()) или, наоборот, которых нет в y (anti_join()).
5.16 Tidy data: tidyr::pivot_longer(), tidyr::pivot_wider()
Принцип tidy data предполагает, что каждая строчка содержит в себе одно измерение, а каждая колонка - одну характеристику. Тем не менее, это не говорит однозначно о том, как именно хранить повторные измерения. Их можно хранить как одну колонку для каждого измерения (широкий формат) и как две колонки: одна колонка - для идентификатора измерения, другая колонка - для записи самого измерения.
Это лучше понять на примере. Например, вес до и после прохождения курса. Как это лучше записать - как два числовых столбца (один испытуемый - одна строка) или же создать отдельную “группирующую” колонку, в которой будет написано время измерения, а в другой - измеренные значения (одно измерение - одна строка)?
- Широкий формат:
| Студент | До курса по R | После курса по R |
|---|---|---|
| Маша | 70 | 63 |
| Рома | 80 | 74 |
| Антонина | 86 | 71 |
- Длинный" формат:
| Студент | Время измерения | Масса (кг) |
|---|---|---|
| Маша | До курса по R | 70 |
| Рома | До курса по R | 80 |
| Антонина | До курса по R | 86 |
| Маша | После курса по R | 63 |
| Рома | После курса по R | 74 |
| Антонина | После курса по R | 71 |
На самом деле, оба варианта приемлимы, оба варианта возможны в реальных данных, а разные функции и статистические пакеты могут требовать от вас как длинный, так и широкий форматы.
Таким образом, нам нужно научиться переводить из широкого формата в длинный и наоборот.
tidyr::pivot_longer(): из широкого в длинный форматtidyr::pivot_wider(): из длинного в широкий формат
new_diet <- tibble(
student = c("Маша", "Рома", "Антонина"),
before_r_course = c(70, 80, 86),
after_r_course = c(63, 74, 71)
)
new_dietТиббл new_diet - это пример широкого формата данных.
Превратим тиббл new_diet длинный:
new_diet %>%
pivot_longer(cols = before_r_course:after_r_course,
names_to = "measurement_time",
values_to = "weight_kg")А теперь обратно в короткий:
new_diet %>%
pivot_longer(cols = before_r_course:after_r_course,
names_to = "measurement_time",
values_to = "weight_kg") %>%
pivot_wider(names_from = "measurement_time",
values_from = "weight_kg")Функция, которая создает другие функции, называется фабрикой функций.↩︎
Как и пакет
tidyverse,tidymodels— это пакет с несколькими пакетами.↩︎Если быть точным, то оператор
%>%был импортирован во все основные пакеты tidyverse, а сам пакетmagrittrне входит в базовый набор tidyverse. Тем не менее, в самомmagrittrесть еще несколько интересных операторов.↩︎Даже наоборот, использование пайпов незначительно снижает скорость выполнения команды.↩︎
Есть споры о том, как это правильно читать. Используемые варианты: диплаер, диплюр, диплир.↩︎
Как и в случае с
magrittr, пакетtidyselectне содержатся в базовом tidyverse, но функции импортируются основыми пакетами tidyverse.↩︎Выбранный паттерн будет найден посимвольно, если же вы хотите искать по регулярным выражениям, то вместо
contains()нужно использоватьmatches().↩︎relocate()не позволяет переименовывать колонки в отличие отselect()иrename()↩︎У функции
dplyr::summarise()есть синонимdplyr::summarize(), которая делает абсолбтно то же самое. Просто потому что в американском английском и британском английском это слово пишется по-разному.↩︎Снять группировку можно с помощью функции
ungroup().↩︎Функция
across()появилась в пакетеdplyrотносительно недавно, до этого для работы с множественными колонками в tidyverse использовались многочисленные функции*_at(),*_if(),*_all(), например,summarise_at(),summarise_if(),summarize_all(). Эти функции до сих пор присутствуют вdplyr, но считаются устаревшими. Другая альтернатива - использование пакетаpurrr(??) или семейства функцийapply()(@ref(apply_f)).↩︎Если ключи будут неуникальными, то функции
*_join()не будут выдавать ошибку. Вместо этого они добавят в итоговую таблицу все возможные пересечения повторяющихся ключей. С этим нужно быть очень осторожным, поэтому рекомендуется, во-первых, проверять уникальность ключей на входе и, во-вторых, проверять тиббл на выходе. Ну или использовать эту особенность работы функции*_join()себе во благо.↩︎